iPhoneのHealthデータをエクスポートしてゴニョゴニョしてみる その3
この記事の続きです。
Google Dataprep登場!
ひとまずiPhoneのHealthデータを取り出すことが出来たので、いよいよNokia Healthのデータと突合してみます。Nokia Healthからの歩数データの記録が抜けている部分をiPhoneのデータで補完をするという話でした。pandasのdataframeでやるのも良いのだけれども、せっかくなので、最近Courseraで学んだGoogle Dataprepを使ってみることにします。

これは、最終的にNokia Healthの歩数データとマージするところまでを含んだデータフローになってます。上のactivities.csvというのがNokia Health側のファイル、export20180520211840.csvというのがiPhoneからのデータの入ったファイル。最後に2本の線が1本にまとまっているのが、2つのファイルをマージするという流れを表しているわけですね。
マージすると言っても、2つのファイルが持っている項目が異なったり、そもそも歩数データ以外の(今は)必要のないデータも入っているので、そこらへんの整理からやる必要がある訳です。まずは、iPhone側のデータから。
レシピを作る
データを加工するにあたって、レシピというのを作り、その中にさらにステップを追加していくというのが基本の流れ。今回作ったレシピには、こんな感じで11ステップ含めてみました。

これだけ見るとウヘーと思うかも知れないけど、やっていることはいらない項目をドロップ(Delete)しているのと、あとは日ごとに集計(Sum)して、さらにいらない行を消したりしている(Keep Rows)ぐらいで、特段難しいことはしていない。Dataprepでは、これらのステップをグラフィカルなUIで簡単に作れるのと、大量データでもきちんと対応出来る(生データは40万件以上ある)のが良いところかなと。
レシピの変更画面に入って、ハマったところなどを書き出しておこうと思います。
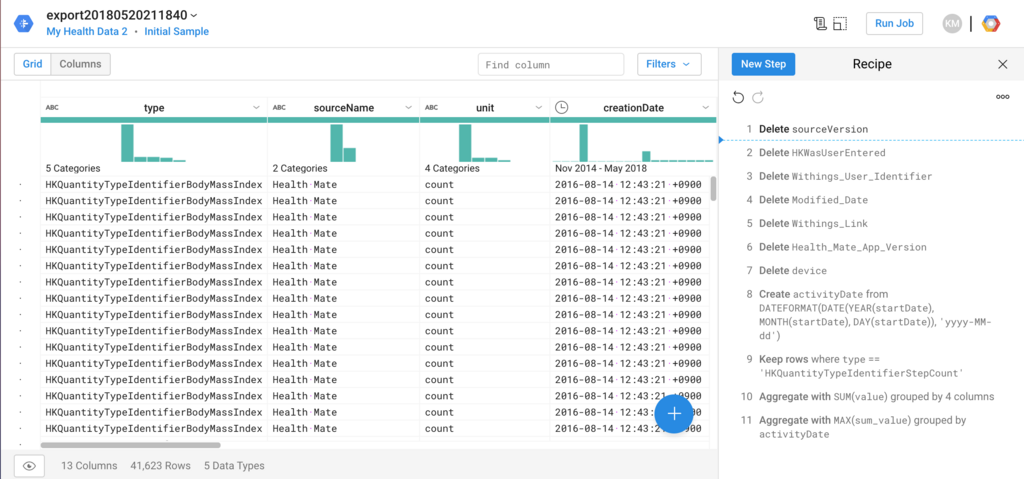
列名とデータの間に分布の棒グラフが出るあたりが、既になんだか良い感じですよね。ここで、1つ注意しないといけないのは、ココに出ている分布だったり、画面の下にある行数というのは、全データではなくて、サンプリングされたデータであるということ。これを見落としてCourseraのテストでなかなかパスできずに痛い目に遭いました。

冷静に見ると分かるのですが、40万件のデータがなぜか4万件程度になっており、自分が使ったデータの場合はだいたい10%位のデータだけが抽出されていたと言う事になります。と言っても、ここに載っていないデータが処理されないという意味ではなく、あくまでこの画面での表示上の話。ちなみに、この画面にあるInitial sampleというリンクをクリックすると、いま抽出されているサンプルの情報が表示されます。例えば、表示されているサンプルが偏りすぎているみたいな場合は、サンプリングをやり直す事が出来ます。ただ、Dataflowのジョブが流れて、それなりに時間が掛かる(そしてリソースを消費するので、わずからながらに課金される)という事はアタマの片隅に入れておいた方が良いと思います。

新しいサンプルを作って選択したところ。画面に表示されているサンプルの内容、値の散らばりが変わっているのが分かる。
長くなったので、続きはまた今度。
(つづく)
iPhoneのHealthのデータをエクスポートしてゴニョゴニョしてみる(その2)
この記事の続きです。
前の記事で、iPhoneのHealthデータをCSVに変換しました。後で本丸であるNokia Health(旧Withings)のデータとマージするんだけど、その前に、もう少しデータの中身を確認しておこうということで、pandasのdataframeの出番ですね。
Healthのデータの中身を見てみよう
>>> import pandas as pd >>> df1 = pd.read_csv('tmp_files/export20180520211840.csv', low_memory=False) >>> df1.describe(include='all')
include='all'オプションを付けたのは、数値項目以外の情報も見たかったから。出てきたのがこちら。(※右端切れてます)
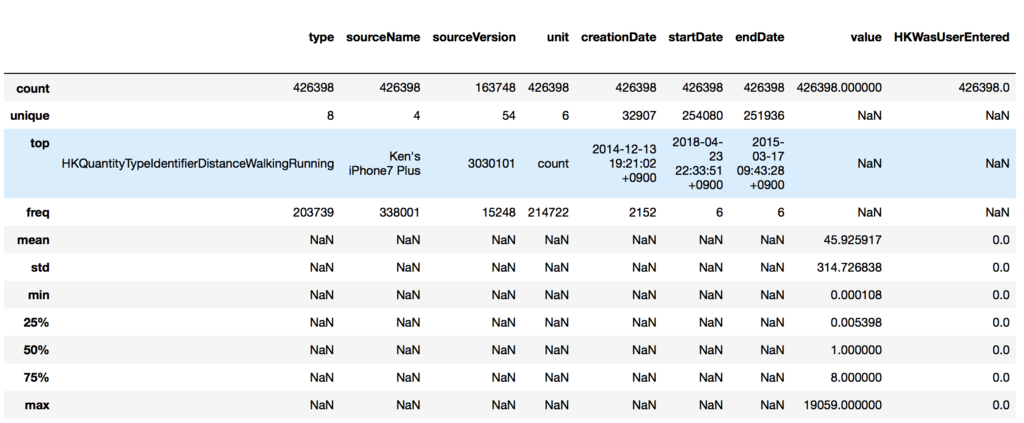
項目ごとに深掘りする必要性があるかを見ていこう。
- type 保持しているレコードの区分のようなものに見える。最頻値「HKQuantityTypeIdentifierDistanceWalkingRunning」は、字面の通りなら歩いたりランニングした距離ということになる。unique値が8ということは、このデータの塊は8種類のデータから出来ているということになる。
- sourceName: 項目名と最頻値から推測すると、データの出元ということになる。iPhone7 Plusからの値が最頻値ではあるが、これもUniqueが4なので4種類のデータがありそう。
- sourceVersion: 何かのバージョンというぐらいしか分からない。全部で42万レコードのうち16万レコードぐらいにしか値が入っていないと読み取れる。Dropしても良いのかな。
- unit: countという如何にも歩数っぽく見える単位が全体の半分を占めている。
- creationDate: レコードが登録された日付と読める。最頻値で2000回以上あるので、一括してインポートした日付なのかなと推定。
- startDate, endDate: 開始・終了の日付。startDateとendDateの最頻値が全く同じに見える。期間値を取ると思っていたので、少し意外。とはいえ、最頻でも6回なので、計測の対象となった時間の情報が入っていると考えるのが正しそう。
- value: これが実際の計測値と思われる。ただ、最小値0.000108で最大値19059なので、全部が全部歩数データではないと言えそう。
- 残りの項目は、MetaAttributeから拾ってきたもの。あまり有益な情報がなさそう。"HKWasUserEntered"は自動入力か手入力かというフラグ、"Withings User Identifier"はユーザID情報に見えるので、あまり重要ではないと判断できそう。ただ、deviceは値が入ったり入っていなかったり。
先頭の数行を出してみる。
>>> df1.head()

カテゴリ値に何があるかを見てみたい。
>>> set(df1.type.values) {'HKQuantityTypeIdentifierActiveEnergyBurned', 'HKQuantityTypeIdentifierBodyFatPercentage', 'HKQuantityTypeIdentifierBodyMass', 'HKQuantityTypeIdentifierBodyMassIndex', 'HKQuantityTypeIdentifierDistanceWalkingRunning', 'HKQuantityTypeIdentifierFlightsClimbed', 'HKQuantityTypeIdentifierHeight', 'HKQuantityTypeIdentifierStepCount'}
歩数が欲しいなら、HKQuantityTypeIdentifierStepCountのデータを拾えと言われている気がする。
>>> set(df1.sourceName.values) {'Health Mate', "Ken's iPhone6", "Ken's iPhone7 Plus", 'UP'}
Health MateはNokiaのアプリの名前、iPhone6, iPhone7 Plusはアプリを経由していない値、UPは自転車乗るときに時々使っていたやつと思われる。
>>> set(df1.unit.values) {'%', 'cm', 'count', 'kcal', 'kg', 'km'}
小数点付きのcountは、どうやらBodyMassIndexのことを指しているんだな。ん?これってBMIのことか!やばい。自分のBMIを公衆の面前に晒してしまった。
・・・ま、7年前のデータだから良いけどね。
歩数関連のデータを深掘りしてみる
先ほど確認したように、歩数関連のデータはtypeの値が'HKQuantityTypeIdentifierStepCount'のものを拾えば良さそう。
>>> df1[df1.type=='HKQuantityTypeIdentifierStepCount'].describe(include='all')

良い感じと思ったものの、value値の散らばりが1~19059というのが気になる。75%値で95なので、19059というのは何かずば抜けた外れ値という可能性もありそう。
となると、10000歩以上記録されているレコードを確認してみよう。
>>> df1[(df1.type=='HKQuantityTypeIdentifierStepCount') & (df1.value>10000)]
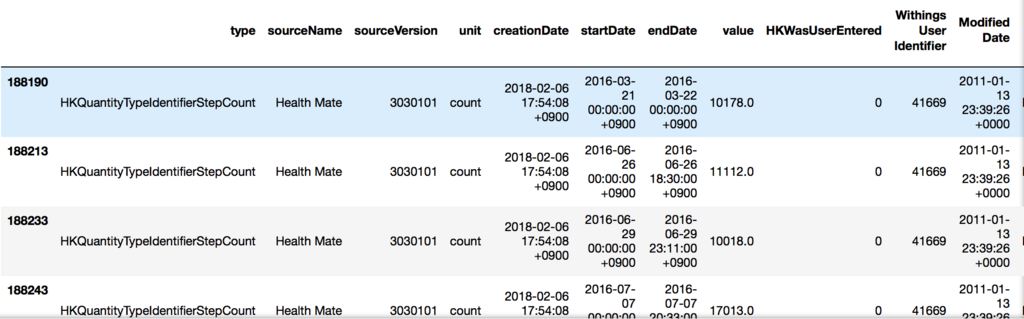
その日の午前0時から夕方または夜までの歩数の合計のように見える。上のイメージでは隠れているけど、全部で141件しかないので、常にこのパターンで記録されているという感じではなさそう。逆に歩数が少ないのは何をやっているのか?
>>> df1[(df1.type=='HKQuantityTypeIdentifierStepCount') & (df1.startDate>"2018-01-15") & (df1.startDate<"2018-01-31")]
※歩数が少ないエントリが2018年アタマに多かったように見えたので、日付で絞っている。
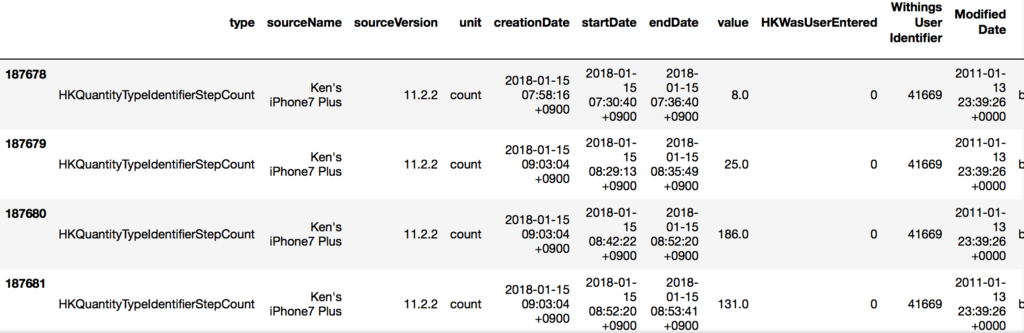
sourceNameがiPhoneになっているのと、startDateとendDateの間が数秒になっている。本来の目的である連携漏れのデータはこちら側に転がっていそうな感じ。
ふむふむ。なんとなく全容が見えた気がするので、Nokia Health側のデータとの突き合わせをやってみよう。
(たぶんつづく)

Pythonデータサイエンスハンドブック ―Jupyter、NumPy、pandas、Matplotlib、scikit-learnを使ったデータ分析、機械学習
- 作者: Jake VanderPlas,菊池彰
- 出版社/メーカー: オライリージャパン
- 発売日: 2018/05/26
- メディア: 単行本(ソフトカバー)
- この商品を含むブログを見る

Pythonによるデータ分析入門 ―NumPy、pandasを使ったデータ処理
- 作者: Wes McKinney,小林儀匡,鈴木宏尚,瀬戸山雅人,滝口開資,野上大介
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/12/26
- メディア: 大型本
- この商品を含むブログ (19件) を見る
iPhoneのHealthデータをエクスポートしてゴニョゴニョしてみる
最近機械学習の流れで、やっぱり自分でこねくり回せるデータの方が色々覚えるかなということで、自分が持っている加工しがいのあるデータを使ってみようかと思い立った週末の午後。と言っても、2010年から取り続けているWithings(現Nokia Health)の体重関連データ、あとは歩数の記録なんかぐらいしか見当たらない。WithingsとiPhoneのHealthデータは基本的に連携していたので、良い感じでデータが残っているものの、なぜか知らぬ間に連携が解除されていた期間があり、そこら辺を補完する意味で、まずはiPhone Healthのデータを解析してみることにしたという顛末記です。
iPhoneのHealthアプリのデータをエクスポートする方法
iOSのバージョンによって多少メニューが異なるもののだいたいこんな感じ。
1. Healthアプリの人影アイコンをタップ

2. 一番下にあるExport Health Dataというメニューを選択

3. 時間かかるけど良い?という確認を無視して、そのままExportをする

4. エクスポートが終わったら、メールに添付するなりしてファイルを入手する
自分の場合は、メールに添付して送信。ZIPファイルの中に2つのXMLファイル(export.xml, export_cds.xml)が入っていたので、ざっくりと中身を確認してみる。まずはサイズの小さそうなexport_cda.xmlから。
import xml.etree.ElementTree as ET with open('tmp_files/export_cda.xml', 'rb') as fd: root = ET.parse(fd).getroot()
どんな情報がありそうかざっくり確認。
>>> root.attrib
{'{http://www.w3.org/2001/XMLSchema-instance}schemaLocation': 'urn:hl7-org:v3 ../../../CDA%20R2/cda-schemas-and-samples/infrastructure/cda/CDA.xsd'}
>>>root.tag
'{urn:hl7-org:v3}ClinicalDocument'
>>>for child in root:
>>> print(child.attrib)
{'code': 'US'}
{'root': '2.16.840.1.113883.1.3', 'extension': 'POCD_HD000040'}
{'root': '2.16.840.1.113883.10.20.22.1.2'}
{'extension': 'Health Export CDA', 'root': '1.1.1.1.1.1.1.1.1'}
{'codeSystem': '2.16.840.1.113883.6.1', 'codeSystemName': 'LOINC', 'code': '34109-9', 'displayName': 'Note'}
{}
{'value': '20180518144502+0900'}
{'code': 'N', 'codeSystem': '2.16.840.1.113883.5.25'}
{}
{}
Clinical documentという時点で、必要としている情報ではなさそう。にしては、ファイルサイズがでかいのが気になるけど、これは必要となったときに戻ってくると言うことにしましょう。ということで、ターゲットはもう1つのexport.xmlにすることに。
export.xmlの中身を確認してみる
同じように、export.xmlの中身を確認してみます。
with open('tmp_files/export.xml', 'rb') as fd: root = ET.parse(fd).getroot()
同じくルートを取ってからの中身の確認。先ほどの手順と同じなので詳細は省きますが、recordというノードの下に欲しそうなデータが転がっている事が判明。
>>> root[2].attrib {'creationDate': '2016-08-14 12:43:21 +0900', 'endDate': '2011-01-14 08:38:33 +0900', 'sourceName': 'Health Mate', 'sourceVersion': '2150100', 'startDate': '2011-01-14 08:38:33 +0900', 'type': 'HKQuantityTypeIdentifierBodyMassIndex', 'unit': 'count', 'value': '31.2459'}
ふむ。では、こんな感じで情報を溜めておけば、
>>> records = [] >>> for child in root: >>> if child.tag == 'Record': >>> records.append(child.attrib)
中身はこうなる。
>>> records[:5] [{'creationDate': '2016-08-14 12:43:21 +0900', 'endDate': '2011-01-14 08:38:33 +0900', 'sourceName': 'Health Mate', 'sourceVersion': '2150100', 'startDate': '2011-01-14 08:38:33 +0900', 'type': 'HKQuantityTypeIdentifierBodyMassIndex', 'unit': 'count', 'value': '31.2459'}, {'creationDate': '2016-08-14 12:43:21 +0900', 'endDate': '2010-08-26 08:17:40 +0900', 'sourceName': 'Health Mate', 'sourceVersion': '2150100', 'startDate': '2010-08-26 08:17:40 +0900', 'type': 'HKQuantityTypeIdentifierBodyMassIndex', 'unit': 'count', 'value': '30.7669'}, {'creationDate': '2016-08-14 12:43:21 +0900', 'endDate': '2010-10-21 08:09:23 +0900', 'sourceName': 'Health Mate', 'sourceVersion': '2150100', 'startDate': '2010-10-21 08:09:23 +0900', 'type': 'HKQuantityTypeIdentifierBodyMassIndex', 'unit': 'count', 'value': '31.1798'}, {'creationDate': '2016-08-14 12:43:21 +0900', 'endDate': '2010-12-10 09:23:06 +0900', 'sourceName': 'Health Mate', 'sourceVersion': '2150100', 'startDate': '2010-12-10 09:23:06 +0900', 'type': 'HKQuantityTypeIdentifierBodyMassIndex', 'unit': 'count', 'value': '31.1798'}, {'creationDate': '2016-08-14 12:43:21 +0900', 'endDate': '2010-03-18 07:24:23 +0900', 'sourceName': 'Health Mate', 'sourceVersion': '2150100', 'startDate': '2010-03-18 07:24:23 +0900', 'type': 'HKQuantityTypeIdentifierBodyMassIndex', 'unit': 'count', 'value': '31.5762'}]
これを基本として、あとでNokia Health側の情報と突き合わせることを考えて、XMLをCSVに変換する事にしました。本当はJSONに変換してゴニョゴニョする予定だったけど、諸事情により中止。この顛末もそのうち記事にするかも。
最終的にCSV変換用に書いたソースがこちら。
Import XML file from Apple Health then convert to ...
長期間のデータが入っているので、アプリのバージョンが変わると、使われている属性(attributes)の顔ぶれが変わっているという問題の対応が必要でした。簡単に言うとアタマのエントリからkeys()を取るだけではダメだった。あとは、数字が入っているはずのvalueという項目になぜか文字列が入っているエントリがあり、最初は項目を変えて残す方向にしていたけど、あまり重要そうではなかったので無視するように変更したあたりがポイントかと。
これで、CSV化が完成したので、今度はこのファイルの中身をゴニョゴニョする感じで行く予定です。
(つづくかも)
Courseraで機械学習+クラウドを学んでみる
連休の暇つぶしに
機械学習本を買い込んで写経をする勉強もいいけど、何か形が残る方が良いかなという事で、CourseraのMachine Learning with TensorFlow on Google Cloud Platformというコースを受講してみることにしました。
このコースを知るきっかけになったのは、このブログです。
この数ヶ月、他の機械学習関連のブログ記事などを見ていると、同じCourseraでもStanfordのMachine Learningがよく紹介されてます。翻訳の方々の活動の賜物で、日本語字幕も用意されており、自分の中でも至れり尽くせりな感じがしていたのは事実ですが、一応これまでの写経生活で積み上げて来たものがあるので、基本的な理論を(またお金を払って)また最初から学ぶのは勿体無いなと思っており、受講を見合わせていました。
そんなタイミングで、CloudベースかつTensorFlowに特化した形でのMLが学べるというオファーはかなり魅力的に映り、連休も近いこともあって試してみるかなという事でジャンプインしてみたわけです。ちなみにお値段は49USD/月。つまり早く終わらせればそれだけ安く上げられるという事ですね。
今のところ、5つあるコースのうち2つを修了して、3つ目のコースの2週目のカリキュラムに取り組んでおり、Courseraでの学びで気がついた事を書き留めておこうと思います。
英語は心配しなくても大丈夫
英語力は人依存じゃんと言われたら身もふたもないのですが、少なくとも英語のスクリプトは画面の下に表示されるので、聞き取りが厳しい人でも読めれば何とかなります。今受けているコースは、講師が入れ替わり立ち代りするのですが、インド風アクセントの人がいたかと思えば、いかにもアメリカン英語な人、多分ロシア系かなてな具合で楽しめます。よく分からなければ、後でスクリプトを見返せば良いので、かなり気楽に構えて大丈夫なはず。
Certificateが貰える
コースによっては、本当に学位が貰えるコースもあるそうですが、今受けているコースでも認証付きの修了証(Certificate)がもらえます。例えば、Linkedinをやっている人であれば、これをそのままAccomplishments のところに貼ることができるので、転職とかを考えている人であれば、最新の技術をキャッチアップしてるよというアピールにもなります。自分の場合もすでに終了したコースのCertificateを2つ貼り付けておきました。
ま、転職活動しているわけではないですけどね…
課題は積極的に取り組むべし
今受講しているコースだと、GCPのコンソールを立ち上げて、DataLabというクラウド上のJupyter Notebookのようなところで課題をこなすようになっているですが、やる気なしモードだと各セルを実行するだけで終わりにできてしまいます。でも、そこで敢えてコードをこねくり回してみたり、追加の練習問題に取り組んでみた方が投資対効果は高いはず。ただ追加の練習問題の解答をどこかに載せて欲しかったなぁ。
と書きつつ、何気に良いなと思ったのが、そこではなくて、時々出てくるDiscussionsというフリーコメントを書くところ。例えば、TensorFlowにおけるVariablesとPlaceholdersの違いを書いてごらんという課題が出てくるんだけど、これが意外に書けない。これの良いところは、他の人がどう書いたかも見れるので、自分の理解は間違ってないなとか、あ、大事なところ外しちゃってるみたいな事が見えてくるんです。自分の言葉でアウトプットして初めて理解できるんだなと気付いてから、まじめに取り組むようにしています。
とひとまずこんなところです。また気づきがあれば共有したいと思います。
機械学習のお勧め本に日本語版が出た!
以前の記事で、機械学習本の中で、一番好きだと言っていたあの本の日本語版が出ました!

scikit-learnとTensorFlowによる実践機械学習
- 作者: Aurélien Géron,下田倫大,長尾高弘
- 出版社/メーカー: オライリージャパン
- 発売日: 2018/04/26
- メディア: 単行本(ソフトカバー)
- この商品を含むブログ (1件) を見る
意外に早いなというのと、英語版と対して値段が変わらないというお得感で、思わずポチッと行ってしまいました。英語版での分かりやすさを損なうことなく、いい感じで日本語化されてて、復習に使えそうです。
ちなみに、同じ日にこの本の日本語版も発売されたので、同じく買ってみました。本当は店頭で確認してから買うつもりだったものの、発売当日に神保町の某大手書店で在庫がないという事態に出くわし、上の本と一緒にポチッてみました。

実践 Deep Learning ―PythonとTensorFlowで学ぶ次世代の機械学習アルゴリズム (オライリー・ジャパン)
- 作者: Nikhil Buduma,太田満久,藤原秀平,牧野聡
- 出版社/メーカー: オライリージャパン
- 発売日: 2018/04/26
- メディア: 単行本(ソフトカバー)
- この商品を含むブログ (1件) を見る
英語版の書評から何となく想像していましたが、最初の2章を読んだ限りでは、入門から入るには少しハードルが高いかなという感じでした。とは言え、この本を手に取る人は、すでに機械学習関連の本を数冊読んでいる可能性が高いので、その知識を動員しながら読めば歯が立たないという事はなさそうです。
昨年末ぐらいに、これからは機械学習の時代だとノリノリだったのですが、これは今籍を置いている会社が機械学習の専業ではないという悲しいところで、思ったほど需要が膨らまず空気的にはややトーンダウンしております。とは言え、生存のために守備範囲は広げておいたほうがいいので、Courseraの機械学習(スタンフォードの有名な方ではなく、Google Cloudの方だけど)などを受講しつつ、来たるべき日に備えております?